Muterat protein
Vid undersökning av ett protein fann man att dess C-terminala del var längre än förväntat. Förlängningen visade sig bero på en mutation i ett exon i den kodande genen. Den bifogade bilden visar C-terminala aminosyrasekvensen i normalt (övre sekvensen) respektive muterat protein (nedre sekvensen).
Vilken typ av mutation har mest sannolikt gett upphov till förlängningen av C-terminalen?
1-baspar insertion
3-baspar insertion
Stoppkodon-deletion
1-baspar substitution
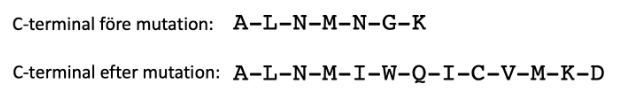
Jag tänkte spontant att det skulle vara 3-baspar insertion eftersom det är nio bokstäver som lagts till utöver de som fanns sedan innan, men detta är fel. Svaret är dock 1-baspar insertion. Hur ska jag tänka?
Nej det är inte nio ytterligare aminosyrarester som lagts till. Om du jämför med den övre korrekta sekvensen, ser du att det de tre sista aminosyraresterna är av en annan typ, samt att sex ytterligare lagts till.
Skulle det vara en 3-baspar insertion, motsvarar det att tre ytterligare baser i DNA lagts till, vilket kan motsvara antingen en ny aminosyrarest (om tripletten hamnar i läsramen "in reading frame"). Alternativt hamnar tripletten ur fas med läsramen, och då fås en förskjutning av läsramen ("out of reading frame").
Att så många som tre extra baspar infogas är mindre sannolikt än att två infogas, och mer sannolikt än två är att endast ett baspar infogas. Det gäller även för deletioner, så att ett helt stoppkodon (3 baser) försvinner är mindre sannolikt.
Och om ett baspar ändras/substitueras påverkas endast den tripletten som basparet ingår i - det kan ge upphov till en förändrad aminosyra i den platsen, men även att ett stopkodon ändras till att koda för en aminosyra t.ex. Trp. Men ofta kodar sekvensen efter stopkodonen inte för riktiga aminosyror (extra stopkodon är också vanliga istället för tripletter som kodar för aminosyror).
Sker det som i svaret en insertion av en ny bas, förskjuts läsramen. T.ex. i DNA före mutationen slutar läsramen med
AAG UAG
Lys Stop
Och tillkommer det en ny bas innan t.ex. ett A förskjuts läsramen
AAA GUG G
Lys Val och så fortsätter det tills ett nytt stopkodon dyker upp.
Du kan m.h.a. en kodontabell se efter vilken typ av 1 baspar insertion som kan ha skett.
Hittar du var i sekvensen insertionen skett?
Med risk för att låta dum förstår jag inte riktigt vad bokstäverna står för. Kvävebaserna som DNA använder sig av är ju A, G, C och T, men jag ser att bokstäverna i sekvenserna verkar baseras på något annat.
Snusmumriken skrev:Med risk för att låta dum förstår jag inte riktigt vad bokstäverna står för. Kvävebaserna som DNA använder sig av är ju A, G, C och T, men jag ser att bokstäverna i sekvenserna verkar baseras på något annat.
Ja det stämmer, det blir ju mRNA när tymin är utbytt till uracil. Mutationen i DNA leder till en muterad sekvens i mRNA, som resulterar i en muterad sekvens i proteinet.
Tillägg: 29 maj 2024 08:15
Jag illustrerade m.a.o. med den proteinkodande sekvensen i mRNA, som får med sig mutationen, som finns i DNA.

